西安交大科研人员在生物信息学软件自适应控制和优化领域取得新突破
得益于新一代基因测序技术和生物信息学的发展,基因组拷贝数变异(CNV)的检测精度日益提高。基因组拷贝数变异是指DNA片段的缺失或重复,是常见的一类基因变异,约占人类基因组的10%以上。CNV与多种遗传性疾病、出生缺陷、癌症等复杂疾病相关,精准检测CNV是临床精准诊疗的关键环节。然而,尽管已有大量基于高通量测序数据的CNV检测软件,在临床检测中仍存在精度低、误差大的问题。究其原因,关键在于基因组的多样性,不同的人在CNV数量、长度等方面均有差别,特别是在疾病样本中此类差异常常较大,而CNV检测软件缺乏对差异的识别和对应的自适应控制和优化能力。因此,面向产业链需求,亟需设计生物信息学软件的自适应控制框架,实现样本级的特异性参数优化。
针对上述问题,西安交通大学计算机科学与技术学院生物信息管理与数字健康研究团队提出了一套通过参数自适应控制实现同时、准确、高效检出广谱长度的拷贝数变异的计算方法。该方法基于样本测序深度概率密度函数的统计控制图,设计了能够划分出序列中不同长度的候选区域的双向快速加权扫描算法,进而针对不同候选区域的特点,由区域内加权深度统计量自适应的设置最优CNV滑窗。滑窗策略与传统CNV检测技术结合,既解决了窗口参数设置过大导致短CNV漏检的问题,又能够避免窗口参数设置过小导致随机扰动难以分辨的问题,实现CNV的精准检出。
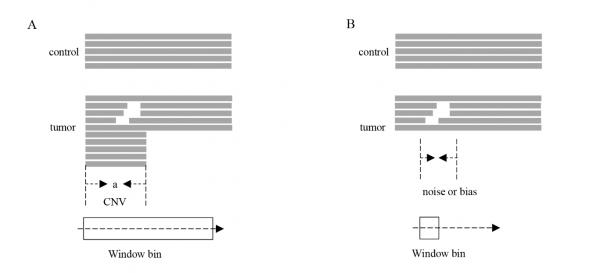
图A 窗口大小对CNV检测的影响
同时,临床常用的肿瘤样本CNV检测场景中,存在肿瘤纯度、肿瘤异质性、肿瘤非整倍性和CNV突变等因素带来的计算复杂性。对此,算法构建了概率模型,利用单个目标碱基周围的碱基的测序深度对目标碱基进行深度矫正。相比于已有的降噪方法,算法在构建待测样本最优基线时避免采集大量额外的样本,更符合临床检测机构的生产实际。
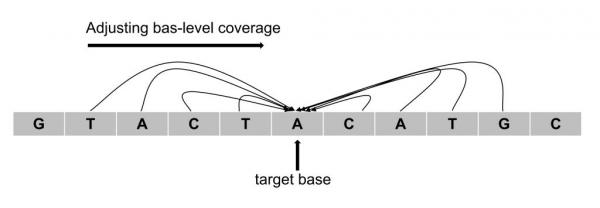
图B目标碱基深度的矫正
以上研究成果近日以《精准、高效地检测不同长度的拷贝数变异》(PEcnv: Accurate and Efficient Detection of Copy Number Variations of Various Lengths)”为题发表在生物信息学领域国际权威期刊《生物信息学简报》(Briefings in Bioinformatics)上。该期刊在数学与计算生物学大类(Mathematical & Computational Biology)的57个期刊中排名第1。生物信息管理与数字健康研究团队王嘉寅教授为论文通讯作者,赖欣副教授、张选平教授、助理教授徐颖、刘玉乾等参与研究工作。
论文链接:https://academic.oup.com/bib/article/23/5/bbac375/6686740
 西安交大科研人员在深度学习辅助核燃料微结构设计方面取得进展
西安交大科研人员在深度学习辅助核燃料微结构设计方面取得进展 西安交大入选第八届恰佩克奖全国产教融合50强
西安交大入选第八届恰佩克奖全国产教融合50强