西安交大科研人员在《自然·方法》发表基因组结构变异检测的突破性研究成果
现有数据表明,基因组结构变异与丰富多彩的生物性状进化和严重疾病表型密切相关。由于结构变异高发区域序列重复度高、存在大量未知复杂类型,传统基于建模策略计算方法存在大量错检、漏检,阻碍深入探究结构变异在生物性状进化和疾病发生中的作用。为了避免由于建模而造成的漏检和错检,谷歌公司首次提出了一种基于深度学习的从图片中直接识别点突变和短插入缺失计算框架,该方法彻底放弃了传统的基于建模的检测方案,同时结合了在大数据背景下人工智能的优势,使得其性能远超业界标准,并迅速在多个领域取代了传统检测方法。然而,面对日益增长的基因组结构变异检测需求,谷歌提出的计算框架只能针对小的插入删除(INDEL)和单核苷酸突变(SNP),并不适用于基因组上大尺度、复杂类型的结构变异。
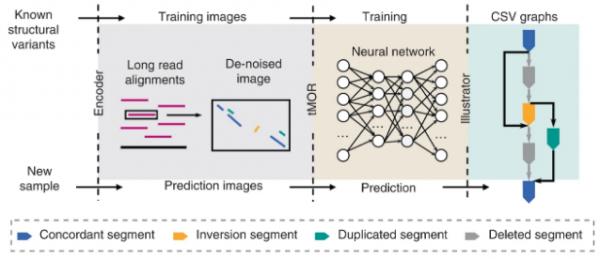
近日,西安交通大学叶凯教授团队在《自然·方法》(Nature Methods)杂志上发表题为《SVision:深度学习方法解析复杂结构变异》(SVision: A deep learning approach to resolve complex structural variants)的研究成果。该研究基于复杂结构变异背景混杂、类型繁多未知的难点,设计了二维序列相似性图,首次将结构变异研究从序列空间建模求解转换为图像空间的多目标识别问题,实现了背景高噪声下未知复杂类型结构变异的精准识别。

该方法主要由编码模块,基于卷积神经网络的多目标识别模块以及基于图结构的复杂结构变异表征模块构成。其中编码模块成功去除了基因组背景噪声对于检测的影响;多目标识别模块实现了只用简单结构变异训练神经网络就可以识别未知复杂类型结构变异的功能,避免了耗费大量资源创建复杂结构变异训练集;复杂结构变异表征模块为后续统一不同研究中报道的复杂结构变异类型提出了重要解决方案,避免了专家导向的复杂结构变异类型定义方式。通过使用该方法,研究者首次系统性地分析了单个人类基因组中携带的复杂结构变异以及其内部结构,揭示了复杂结构变异在影响基因功能和基因组进化中起到的重要作用。本项工作填补了领域内复杂结构变异检测及表征的方法空白,为后续大规模人群队列全类型结构变异研究提供了新的解决方案。
西安交通大学自动化学院博士生蔺佳栋和王松渤为该论文共同第一作者,叶凯教授为唯一通讯作者。
近年来,叶凯教授致力于交叉团队建设以及探索交叉学科创新合作模式,本项目成果就得益于自动化、数学以及生物学的交叉创新,此项目也为后续交叉合作提供了宝贵经验。
文章链接地址:https://www.nature.com/articles/s41592-022-01609-w
 国家重点研发计划“页岩油开发可控冲击波压裂技术的基础研究”项目中期检查会召开
国家重点研发计划“页岩油开发可控冲击波压裂技术的基础研究”项目中期检查会召开 物理学院组织观看“庆祝教师节暨表彰大会”并召开新入职教师座谈会
物理学院组织观看“庆祝教师节暨表彰大会”并召开新入职教师座谈会